V. Nägerl, J.B. Sibarita et al dans Nature Methods
Commentaire
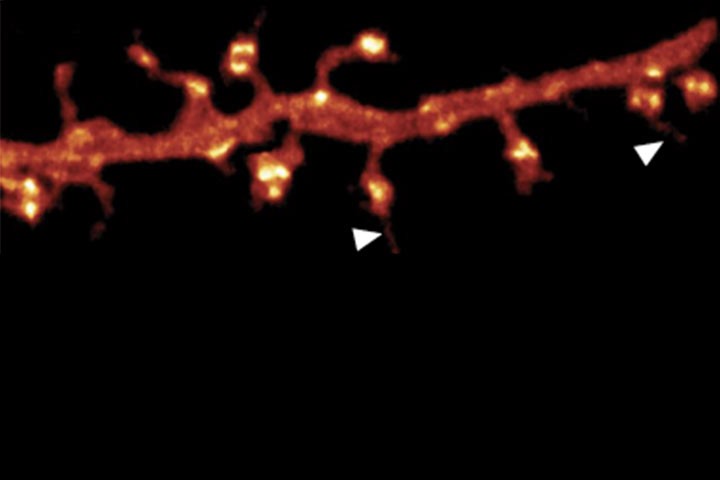
La microscopie de super-résolution offre d’énormes possibilités de démêler l’architecture complexe et dynamique des cellules vivantes. Cependant, les microscopes de super-résolution actuels sont bien adaptés pour révéler la distribution des protéines ou la morphologie cellulaire, mais pas les deux. Les chercheurs ont surmonté ce problème en combinant l’imagerie de molécules uniques et la microscopie STED sur une seule plateforme, permettant de révéler la morphologie et l’organisation moléculaire des synapses à des échelles nanoscopiques. L’étude a été publiée dans la revue Nature Methods.
Voir le commentaire complet sur le site web du CNRS
Abstract
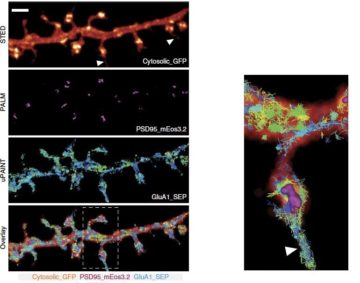
Super-resolution microscopy offers tremendous opportunities to unravel the complex and dynamic architecture of living cells. However, current super-resolution microscopes are well suited for revealing protein distributions or cell morphology, but not both. We present a super-resolution platform that permits correlative single-molecule imaging and stimulated emission depletion microscopy in live cells. It gives nanoscale access to the positions and movements of synaptic proteins within the morphological context of growth cones and dendritic spines.
https://www.nature.com/articles/s41592-019-0611-8
Contacts:
Jean-Baptiste Sibarita & Valentin Nägerl, IINS
Mise à jour: 13/11/19
