Un logiciel pour mieux analyser les données de super-résolution
La microscopie de super-résolution par localisation de molécules individuelles, couronnée par le prix Nobel de chimie 2014, est une technique de microscopie permettant de sonder l’intimité cellulaire avec des résolutions quasi-nanométriques. Adoptée par de nombreux laboratoires de biologie, elle reste encore sous-exploitée, notamment à cause de la nature pointilliste de ses données. Un nouveau logiciel, développé par Florian Levet (BIC) et Jean-Baptiste Sibarita (IINS), et publié dans la revue Nature Methods, offre la possibilité de manipuler et quantifier efficacement ces données.
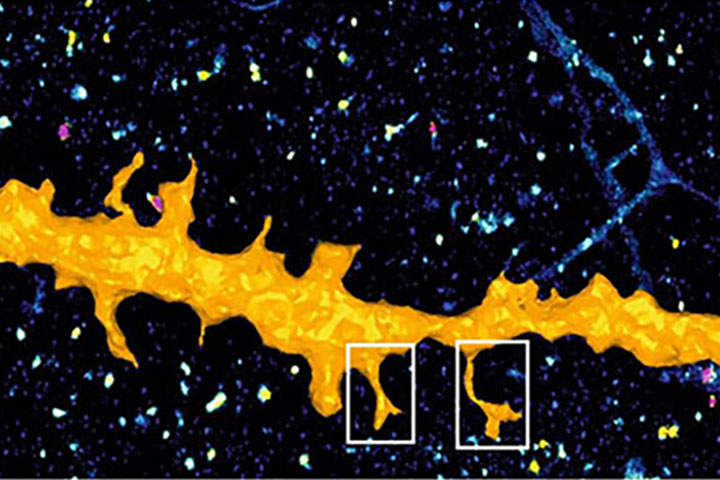
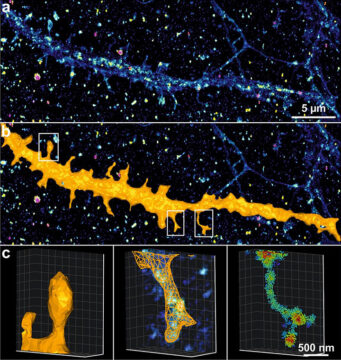
La microscopie de super-résolution par localisation de molécules individuelles, ou SRLMI, permet de reconstruire une image à partir de l’ensemble des coordonnées de fluorophores. Pour ce faire, la stratégie consiste à n’activer qu’une très faible fraction des fluorophores présents dans l’échantillon, afin de les isoler spatialement et ensuite pouvoir les localiser informatiquement avec des précisions pouvant atteindre quelques nanomètres. L’image finale, dite de super-résolution, est alors reconstruite à partir de l’ensemble des coordonnées des molécules extraites à partir de milliers d’images successives collectées et analysées.
Initialement réservée aux experts en microscopie et analyse d’images, la microscopie SRLMI s’est rapidement démocratisée, permettant de réaliser des avancées majeures en biologie cellulaire et neurosciences. Elle est aujourd’hui utilisée en routine, offrant aux scientifiques la capacité d’étudier l’organisation spatiale de protéines à l’intérieur de cellules à l’échelle nanométrique, résolution jusqu’alors uniquement accessible grâce à la microscopie électronique. Elle permet également de suivre précisément la dynamique de molécules avec des résolutions temporelles de l’ordre de la milliseconde, alors que la microscopie électronique n’autorise pas l’observation d’échantillons vivants.
Malgré tout, cette technique de microscopie reste encore sous-exploitée, notamment à cause de la nature pointilliste et de la quantité des données qu’elle génère. En effet, les images de super-résolution peuvent contenir jusqu’à plusieurs millions de coordonnées de molécules en 3 dimensions, rendant leurs manipulations et leurs interprétations complexes. Ainsi, la majorité des solutions logicielles reconstruisent des images d’intensité à partir des coordonnées de localisation, permettant d’utiliser les outils de visualisation et d’analyse d’images traditionnels. Néanmoins faire cette opération induit une perte d’information considérable, à savoir les coordonnées spatiales et temporelles de chaque protéine marquée et localisée.
Ces dernières années, les scientifiques ont travaillé sur le développement de nouvelles méthodes d’analyse permettant d’exploiter au mieux la nature pointilliste des données de localisation. Néanmoins, ces méthodes ne sont pas toujours accessibles à des non-experts, ni optimisées lorsqu’il s’agit de traiter des données expérimentales constituées d’un nombre important de localisations. Ce constat illustre un manque flagrant d’outils polyvalents et optimisés permettant d’intégrer un maximum de méthodes d’analyses compatibles avec les données de localisation 2D, 3D et multi-couleurs.
Pour pallier à ce manque, les scientifiques ont développé PoCA (Point Cloud Analyst), un nouveau logiciel permettant l’analyse multidimensionnelle de nuages de points acquis en SRLMI. PoCA intègre un moteur de visualisation 3D avancé, des capacités de manipulation et d’interaction performantes, ainsi que des techniques de quantification de référence, rapides et robustes. Héritier des deux précédents logiciels SR-TESSELER et COLOC-TESSELER, PoCA est basé sur une architecture acceptant les « plugins », permettant ainsi l’intégration de méthodes existantes et futures d’analyse de données de localisation. Il intègre également un interpréteur Python et un moteur d’exécution de macros, facilitant le prototypage de nouvelles méthodes et la reproductibilité des analyses.
Référence
PoCA: a software platform for point cloud data visualization and quantification.
Florien Levet, Jean-Baptiste Sibarita
Nat Methods (2023).
DOI : https://doi.org/10.1038/s41592-023-01811-4
Contact
Jean-Baptiste Sibarita
Ingénieur de recherche CNRS, IINS
Mise à jour: 10/05/23
