soSMARt : Sonder le vivant à l’échelle nanométrique en profondeur
Rémi Galland et Jean-Baptiste Sibarita publient dans Nature Communications une nouvelle méthode d’imagerie de super-résolution, baptisée soSMARt, qui permet d’explorer l’organisation nanométrique de protéines dans des cellules entières et des cultures multi-cellulaire 3D. En combinant une technique d’imagerie par feuille de lumière développée au laboratoire avec de nouveaux dispositifs d’imagerie, cette approche ouvre la voie à une exploration plus profonde et précise du vivant dans des échantillons 3D complexes.
Description
Comprendre l’organisation des protéines à l’échelle nanométrique dans leur environnement cellulaire naturel est un enjeu central pour de nombreuses recherches en biologie. La microscopie de localisation de molécules uniques (SMLM), une technique d’imagerie dite « super-résolue », permet depuis une vingtaine d’années de repousser les limites de résolution de la microscopie optique conventionnelle. Toutefois, cette technologie reste majoritairement restreinte à l’imagerie de zones proches de la lamelle, limitant ainsi l’exploration de l’intérieur des cellules ou des tissus.
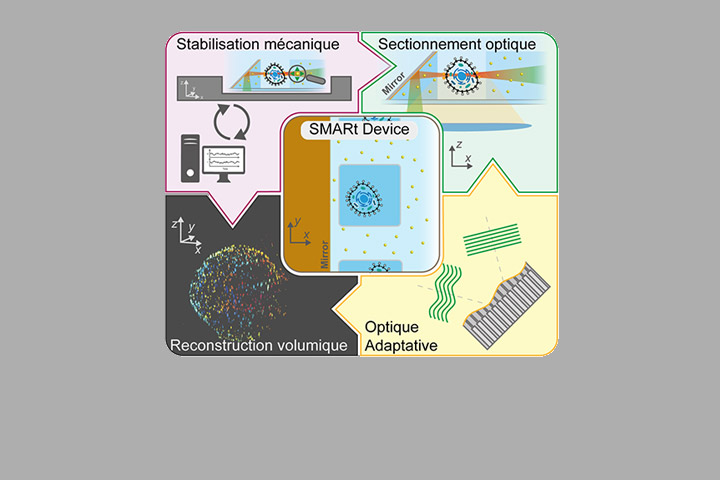
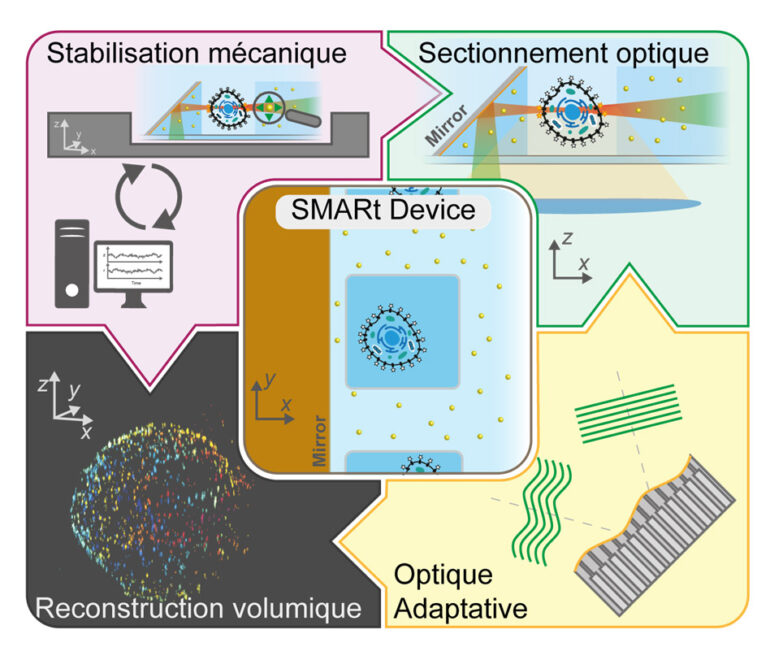
Dans ce contexte, nous avons développé la plateforme d’imagerie soSMARt (single-objective Single Molecule Active Registration technique), une méthode permettant pour la première fois de combiner imagerie en profondeur, super-résolution spatiale, stabilité temporelle sur plusieurs heures, et acquisition automatisée de volumes 3D entiers.
La clé de cette avancée réside dans l’utilisation de dispositifs microfabriqués innovants, appelés « SMARt devices », intégrant :
- Des miroirs inclinés à 45°, permettant une illumination par feuille de lumière de l’échantillon tout en maximisant la collection du signal de fluorescence (technologie soSPIM développé au sein de l’équipe)
- Des marqueurs fluorescents stables servant de références pour (i) la correction des dérives mécaniques en temps réel, (ii) la correction des aberrations optiques par Optique Adaptative (OA), et (iii) la reconstruction de volumes entiers avec des résolutions nanométriques
Enfin, un logiciel de pilotage (SMARtrack) assure la stabilisation active du système et l’automatisation de l’acquisition sur des volumes d’environ 20x20x20 µm3. Grâce à cette combinaison, soSMARt permet d’imager des cellules entières ou des agrégats cellulaires plusieurs dizaines de microns en profondeurs avec une résolution spatiale pouvant atteindre 7 nm latéralement et 40 nm axialement.
Nous avons validé cette approche sur différents systèmes biologiques : de cellules adhérentes, à des cellules immunitaires en suspension, et des sphéroïdes tumoraux en 3D, démontrant la capacité de cette plateforme d’imagerie à cartographier l’organisation 3D de protéines clés en profondeur, comme les récepteurs membranaires PD-1 ou CD3 d’intérêt en immunothérapie. Enfin, l’intégration d’approches d’intelligence artificielle (deep learning) a permis d’accélérer le processus d’acquisition d’un facteur 10 et d’améliorer la qualité de reconstruction des volumes super-résolues.
Avec la plateforme soSMARt, la microscopie super-résolue par localisation de molécules individuelles devient compatible avec l’imagerie de cellules entières, et ouvre la voie à l’observation de l’organisation nanométriques de protéines à l’intérieurs d’échantillons 3D complexe (Sphéroïdes, Organoïdes) plus représentatifs des conditions physiologiques d’organes réels.
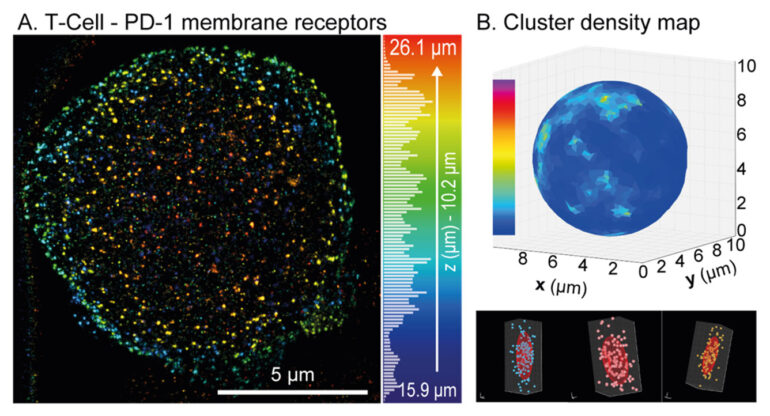
Figure : (A) Reconstruction 3D de la répartition des récepteurs PD-1 sur l’ensemble de la membrane d’une cellule immunitaire T suspendue à l’intérieur d’un dispositif SMARt, la couleur codant pour la profondeur. (B) Projection sur une sphère de la carte de densité des clusters formés par les récepteurs PD-1 (haut), avec trois exemples de ces clusters (bas).
Référence
Cabillic, H. Forriere, L. Bettarel, C. Butler, A. Neuhaus, I. Idrissi, M.E. Sambrano-Lopez, J. Rossbroich, L-R. Müller, J. Ries, G. Grenci, V. Viasnoff, F. Levet, J-B. Sibarita & R. Galland, “In-depth single molecule localization microscopy using adaptive optics and single objective light-sheet microscopy”, Nature Communications 16, 8362 (2025)
https://doi.org/10.1038/s41467-025-62198-8
Contact
Rémi Galland
Chargé de Recherche CNRS
Institut Interdisciplinaire de Neurosciences (IINS)
CNRS / Université de Bordeaux
Centre Broca Nouvelle Aquitaine
146 rue Léo Saignat
CS 61292 Case 130
F-33076 Bordeaux Cedex (France)
Mise à jour: 14/10/25
